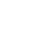新方法实现多倍体准确基因分型
时间:2021-07-25 来源:
Molecular Ecology Resources 新方法实现多倍体准确基因分型、克服多倍体种群遗传多样性分析基础难题:以六倍体油茶为例
近日,南昌大学流域生态学研究所、天昊生物、中国科学院庐山植物园和复旦大学研究人员联合在著名生态学期刊Molecular Ecology Resources发表了题为“High-throughput sequencing-based microsatellite genotyping for polyploids to resolve allele dosage uncertainty and improve analyses of genetic diversity, structure and differentiation: A case study of the hexaploid Camellia oleifera”的研究论文(Cui et al. 2021)。该研究以六倍体油茶为例,提出了新方法实现多倍体准确基因分型,克服了多倍体种群遗传多样性分析的基础难题。

微进化就是等位基因频率的变化。准确估计等位基因频率是关键,是遗传多样性、遗传结构和遗传分化分析的基础。常规的基因分型技术和群体遗传学分析方法主要针对二倍体,可用分子标记对二倍体样本准确基因分型,计算群体中的等位基因频率,进而获得可靠的种群遗传多样性、遗传结构和遗传分化分析结果。但是,针对多倍体的基因分型技术和群体遗传学分析方法还不成熟。主要的挑战是如何解决多倍体等位基因数量不确定(allele dosage uncertainty)的难题(图1)。

图1 常规方法难以对多倍体进行准确的基因分型,导致等位基因数量不确定(allele dosage uncertainty)。
多倍化在被子植物多样化中起重要作用。约15%的被子植物物种形成事件伴随着倍性增加,约35%的被子植物物种是多倍体(Wood et al. 2009)。由于多倍体常比二倍体近缘种长得更大、更快并具有更高产量,多倍化也可以促进作物的驯化和改良。许多重要作物是多倍体,如:四倍体土豆(Solanum tuberosum)、六倍体小麦(Triticum aestivum)和四倍体油菜(Brassica napus)。因此,对多倍体进行准确的基因分型和群体遗传学分析,对于了解被子植物的演化、作物驯化和遗传改良均具有重要意义。
微卫星或简单序列重复(SSRs)是群体遗传学研究中最流行的分子标记之一。常规的微卫星基因分型技术无法准确鉴别多倍体的基因型,导致等位基因数量不确定。因此,在多倍体中,共显性的微卫星基因型常不得不以类似显性分子标记数据的处理方式进行分析,从而丢失了大量的等位基因及频率信息。另一方面,类似GenoDive的少数软件能处理等位基因数量不确定的微卫星基因型数据,可用最大似然法对等位基因的数量进行校正(假设种群内随机交配)。由于实际的等位基因频率未知,这类校正会使种群遗传分化和遗传结构分析产生偏差。
有一些方法基于毛细管电泳中微卫星等位基因的峰面积比例来推测多倍体基因型。但是,毛细管电泳中微卫星等位基因的峰面积比例可能不反映实际的等位基因比例,特别是在没有考虑微卫星等位基因的滑移峰(stutter peak)和扩增效率问题的情况下。微卫星的滑移峰会被误判成等位基因峰,或与等位基因峰重叠使峰面积比例产生偏差;等位基因片段长度越长(微卫星重复次数越多),扩增效率往往越低,也会使峰面积比例产生偏差。这均会影响等位基因数量的准确估计。
Meirmans et al.(2018)通过模型模拟显示,如果等位基因数量不确定,多倍体遗传多样性分析会产生明显偏差:四倍体种群的观测杂合度会远高于真实值,而预期杂合度则略高于真值,并且无法进行哈迪-温伯格平衡的检验。即使等位基因数量不确定,Stift et al.(2019)用模拟显示仍可用STRUCTURE软件对多倍体种群进行遗传结构分析。缺少等位基因数量信息会高估多倍体种群的遗传多样性从而低估种群间的遗传分化水平,模拟显示经典的遗传分化系数Fst可能难以反映多倍体种群间的遗传分化(Meirmans & van Tienderen 2013; Meirmans et al. 2018)。
亟需开发新的方法实现准确的多倍体基因分型,才能从根本上解决多倍体种群遗传多样性分析的基础难题。Cui et al.(2021)研究开发了新的基于高通量测序的微卫星基因分型方法(图2)用于解决多倍体基因分型中等位基因数量不确定的难题。

图2 新的基于高通量测序的微卫星基因分型技术路线(修改自Cui et al. 2021)。SSRSeq count是Perl脚本把测序数据转换成微卫星序列数量作为后续分析的输入数据。SSRSeq V1.1软件计算微卫星序列的频率分布,鉴别微卫星等位基因,进行滑移峰校正和扩增效率校正,获得校正等位基因数量的微卫星基因型。
山茶属(Camellia)中存在不少的多倍体,特别是在油茶组(Paracamellia)中。油茶(Camellia oleifera)是油茶组的模式物种,为常绿阔叶灌木或小乔木,主要是六倍体(图3)。栽培油茶是我国第一大木本油料作物。油茶籽油富含单不饱和脂肪酸——油酸(高达80%以上),被誉为“东方橄榄油”,是优质健康的食用植物油。野生油茶(C. camellia)是栽培油茶育种宝贵的遗传资源,广泛分布于长江流域及其以南的亚热带常绿阔叶林中。了解野生油茶种群遗传多样性的空间分布格局是野生遗传资源挖掘与利用的基础。


图3 油茶花和果实
Huang et al.(2018)用8个微卫星分子标记以常规毛细管电泳对庐山和井冈山野生油茶进行了基因分型。遗传多样性分析显示观测杂合度显著大于预期杂合度;遗传结构分析提示庐山和井冈山野生油茶间存在明显的遗传分化,每座山内不同海拔间的遗传分化较小。然而,经典的遗传分化系数Fst显示,庐山和井冈山间的遗传分化极低(Fst = 0.007),与每座山内的遗传分化相等。该研究中野生油茶均为六倍体,常规基因分型导致的等位基因数量不确定会使等位基因频率的估计产生明显偏差,从而可能影响遗传多样性分析结果的可靠性。
Cui et al.(2021)采用新的基于高通量测序的微卫星基因分型方法(图2),用35个微卫星分子标记对六倍体野生油茶种群样本进行了基因分型。结果显示,微卫星的滑移比例(重复次数为n − 1的滑移序列频率/重复次数为n的微卫星序列频率)随微卫星重复次数的增加而增加(图4)。该研究开发了基于滑移比例的校正方法进行滑移峰校正,以获得更为准确的微卫星基因分型结果。此外,微卫星扩增比例(观测等位基因数量/预期等位基因数量)随微卫星重复次数的增加有下降的趋势(图5)。该研究开发了基于扩增比例的校正方法进行扩增效率校正,以获得更为准确的微卫星基因分型结果。该研究提供了SSRSeq V1.1软件进行相应的校正,最后输出校正等位基因数量的微卫星基因型。六倍体野生油茶种群样本的研究结果显示,校正的等位基因数量与预期的等位基因数量高度一致,说明新的方法可以获得准确的多倍体基因型。
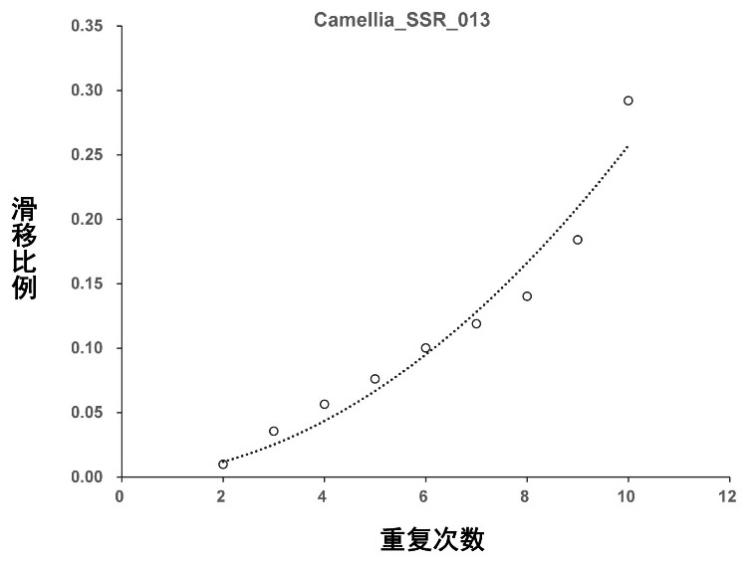
图4 微卫星滑移比例与重复次数的相关性(修改自Cui et al. 2021)。仅显示微卫星分子标记Camellia_SSR_013在六倍体野生油茶样本中的扩增测序结果(完整的结果详见Cui et al. 2021)。
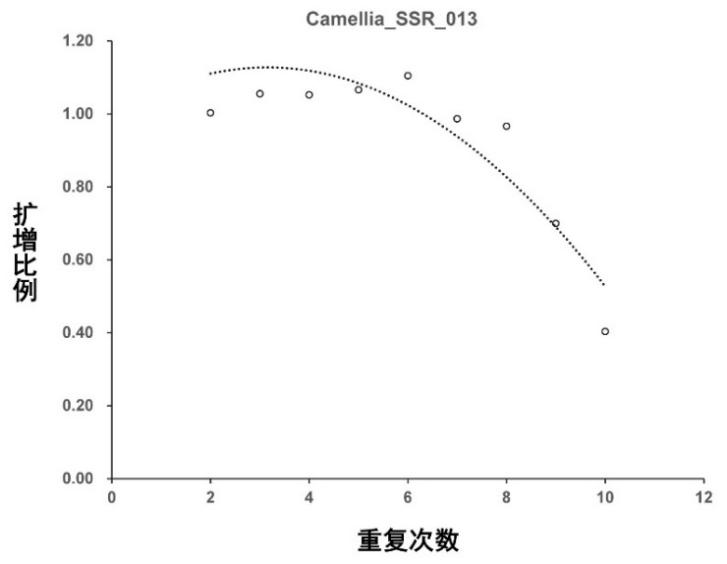
图5 微卫星扩增比例与重复次数的相关性(修改自Cui et al. 2021)。仅显示微卫星分子标记Camellia_SSR_013在六倍体野生油茶样本中的扩增测序结果(完整的结果详见Cui et al. 2021)。
Cui et al.(2021)比较了校正等位基因数量(等位基因数量确定)与未校正等位基因数量(等位基因数量不确定)的情况下,六倍体野生油茶种群遗传多样性、遗传结构和遗传分化分析的差异。该研究结果显示,是否校正等位基因数量会对分析结果造成明显影响。
Cui et al.(2021)发现,校正等位基因数量后,六倍体野生油茶种群的观测杂合度(< 0.6)均显著低于预期杂合度,近交系数为正;未校正等位基因数量,观测杂合度均异常高(> 0.8),显著高于预期杂合度,近交系数为负(图6)。该结果与Meirmans et al.(2018)模型模拟结果类似,说明未校正等位基因数量会严重高估六倍体野生油茶种群的杂合度,尤其是观测杂合度,导致偏离实际情况的统计推断。野生油茶自交不亲和,通过昆虫传粉异交,种子通过小型啮齿动物在林下传播,种群内基因扩散距离有限,因此种群的观测杂合度应显著低于预期杂合度,这与校正等位基因数量的结果一致。

图6 六倍体野生油茶种群的观测和预期杂合度(修改自Cui et al. 2021)。(a)校正等位基因数量;(b)未校正等位基因数量。
Cui et al.(2021)发现,是否校正等位基因数量均可揭示六倍体野生油茶种群中明显的遗传结构,如:庐山野生油茶种群(LU)具有独特的遗传结构,与其他种群的遗传分化最明显(图7和图8)。庐山地处中亚热带与北亚热带交界区,位于野生油茶分布区北部,分布有耐低温胁迫的野生油茶资源;庐山北临长江、东南与鄱阳湖为邻,与其他野生油茶种群分布地具有明显的地理隔离。低温等气候条件的适应性隔离以及地理隔离可能共同造成庐山野生油茶种群独特的遗传结构。但是,校正等位基因数量可以揭示更为精细的种群遗传结构(Cui et al. 2021),如:位于野生油茶分布区南部、地处南亚热带的罗浮山野生油茶种群(LF)此时显示出明显的遗传分化(图7和图8)。
图7 六倍体野生油茶种群样本PCA分析结果(修改自Cui et al. 2021)。(a)校正等位基因数量;(b)未校正等位基因数量。
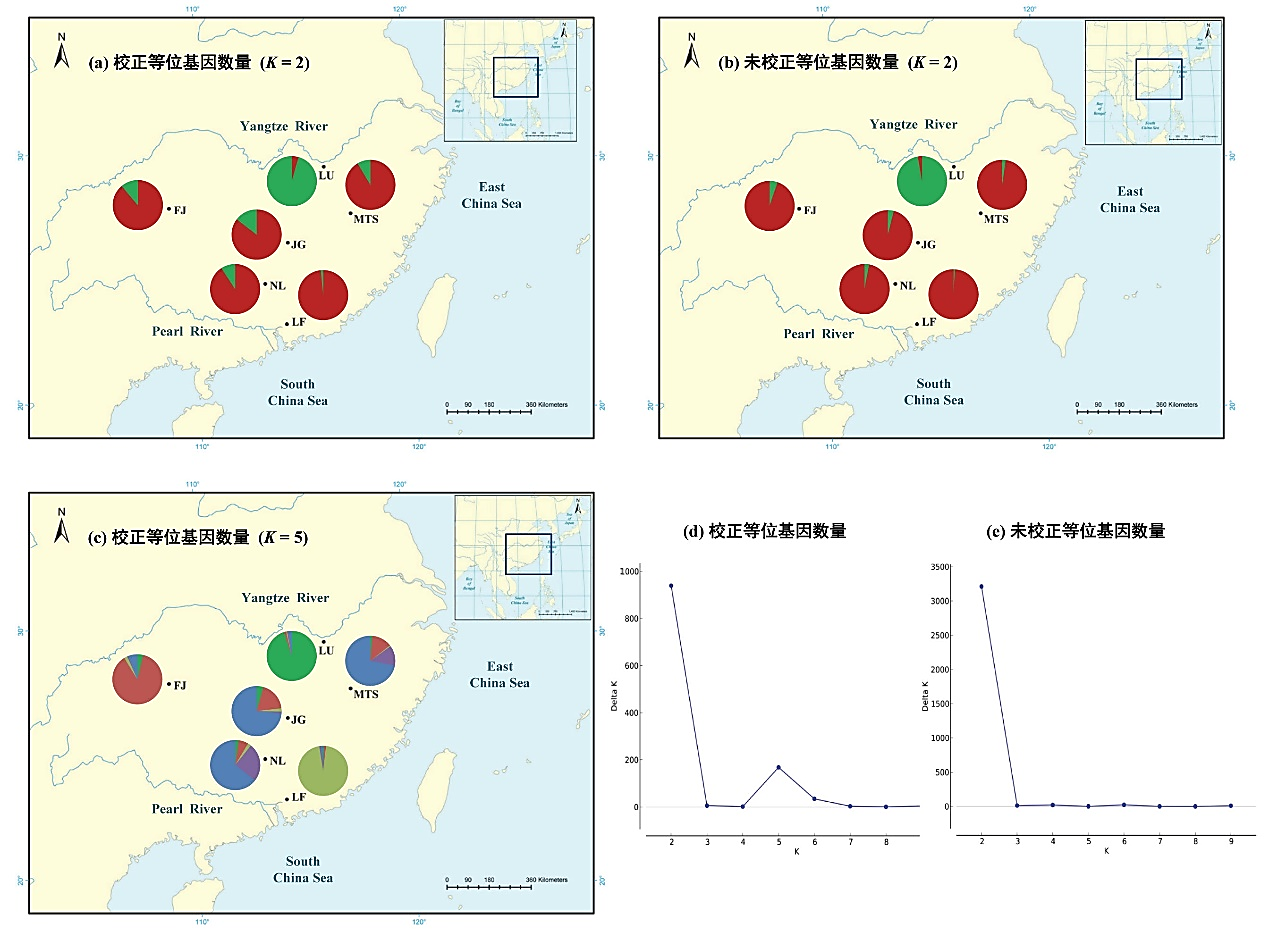
图8 六倍体野生油茶种群遗传结构(修改自Cui et al. 2021)。(a)校正等位基因数量(K = 2);(b)未校正等位基因数量(K = 2);(c)校正等位基因数量(K = 5);(d)校正等位基因数量的∆K分析结果;(e)未校正等位基因数量的∆K分析结果。
Cui et al.(2021)发现,与Meirmans et al.(2018)模型模拟结果类似,未校正等位基因数量会低估六倍体野生油茶种群间的遗传分化,导致极低的Fst估计。校正等位基因数量后,Fst值可以反映与种群遗传结构类似的遗传分化格局(Cui et al. 2021)。
综上所述,Cui et al.(2021)通过六倍体野生油茶种群的研究证明,未校正等位基因数量会对多倍体种群遗传多样性、遗传结构和遗传分化分析结果造成严重偏差。该研究开发的基于高通量测序的微卫星分型新方法可以准确估计多倍体等位基因数量,从根本上解决困扰多倍体种群遗传多样性、遗传结构和遗传分化分析的基础难题。相关的分析软件SSRSeq V1.1(http://bioinfo.geneskybiotech.com/software/ssrseq_type/v1.1/)可以输出微卫星等位基因鉴别、滑移校正、扩增效率校正等中间结果,方便用户根据具体情况选择合适的分子标记和调整运行参数;输出的微卫星基因型(GenoDive格式)包括校正等位基因数量和未校正等位基因数量的结果,方便对两者分别进行分析和比较。
该研究由南昌大学流域生态学研究所、天昊生物、中国科学院庐山植物园和复旦大学的研究人员合作完成。南昌大学流域生态学研究所硕士崔相艳和天昊生物生物信息部李才华是论文的共同第一作者,南昌大学流域生态学研究所和中国科学院庐山植物园戎俊研究员和赵耀副研究员为共同通讯作者。共同作者还包括南昌大学流域生态学研究所秦声远、黄小毛、杨小强和向小果研究员,天昊生物黄泽斌、甘斌和姜正文,以及复旦大学李琴和陈家宽教授。该研究得到国家重点研发计划项目(2018YFD1000603)、国家自然科学基金面上项目(31870311)和江西省人才项目的资助。
主要参考文献
Cui XY, Li CH, Qin SY, Huang ZB, Gan B, Jiang ZW, Huang XM, Yang XQ, Li Q, Xiang XG, Chen JK, Zhao Y, Rong J. 2021. High-throughput sequencing-based microsatellite genotyping for polyploids to resolve allele dosage uncertainty and improve analyses of genetic diversity, structure and differentiation: A case study of the hexaploid Camellia oleifera. Molecular Ecology Resources. https://doi.org/10.1111/1755-0998.13469.
Huang XM, Chen JM, Yang XQ, et al. 2018. Low genetic differentiation among altitudes in wild Camellia oleifera, a subtropical evergreen hexaploid plant. Tree Genetics & Genomes 14, 21.
Meirmans PG, Liu S, van Tienderen PH. 2018. The Analysis of Polyploid Genetic Data. Journal of Heredity 109, 283-296.
Meirmans PG, van Tienderen PH. 2013. The effects of inheritance in tetraploids on genetic diversity and population divergence. Heredity 110, 131-137.
Stift M, Kolář F, Meirmans PG. 2019. STRUCTURE is more robust than other clustering methods in simulated mixed-ploidy populations. Heredity 123, 429-441.
Wood TE, Takebayashi N, Barker MS, et al. 2009. The frequency of polyploid speciation in vascular plants. Proceedings of the National Academy of Sciences of the United States of America 106, 13875-13879.